【90】次世代シークエンサー
1977年にフレデリック・サンガーによって、DNAの塩基配列を読み取る方法(サンガー法/ジデオキシ法)が開発された。
1990年に始まった〈ヒトゲノム計画〉では、ヒトの核DNAの全塩基配列を、15年程かけて読み取る計画だったが、塩基配列を自動で読み取る装置(自動シークエンサー)の実用化などにより、予定より2年早い2003年までに作業が完了した。
その後もDNAの塩基配列読み取り方法(DNAシークエンス)は改良され続け、現在では、ヒトゲノム(30億塩基対)の塩基配列を数日で読み取ることができるようになった。
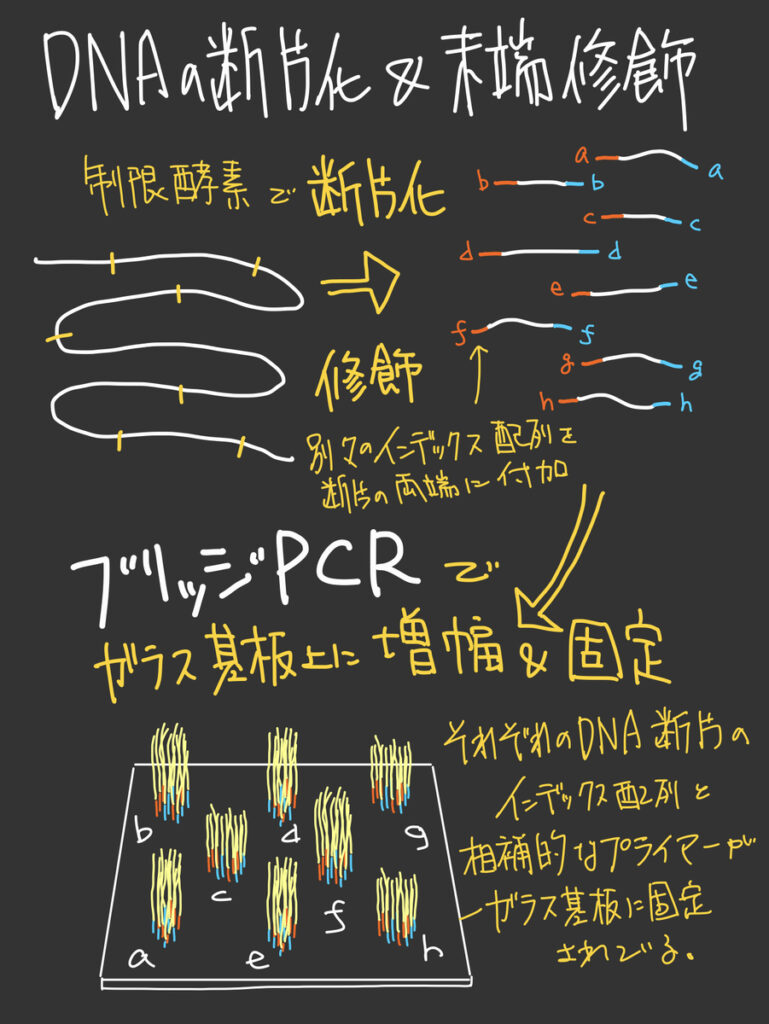
ガラス基板の場所ごとに、それぞれのDNA断片のインデックス配列(末端部)に相補的なプライマーが、5’末端をガラス基板に固定された状態で固定されている。

ガラス基板上の場所ごとに、別々のDNA断片が増幅されるので、同時に複数のDNA断片の塩基配列を読み取ることができる。
〈次世代シーケンス〉にも様々な方法があるが、どの方法も”同時に複数のDNA断片の塩基配列を読み取れることで、解析速度が増している。
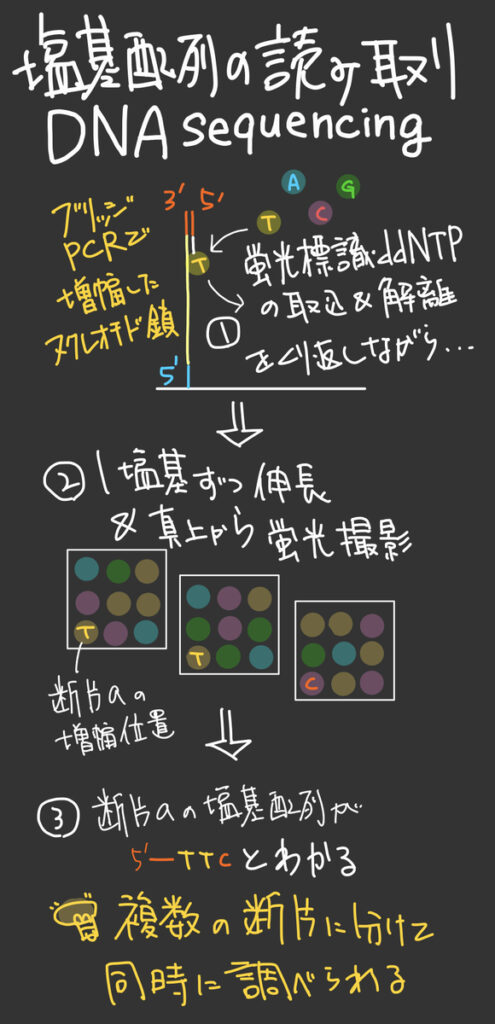
〈次世代シーケンス/次世代シーケンサー〉で、大量のDNAの塩基配列をより速く読み取れるようになったことで、SNPなどの個人差を比較することもできるようになった。
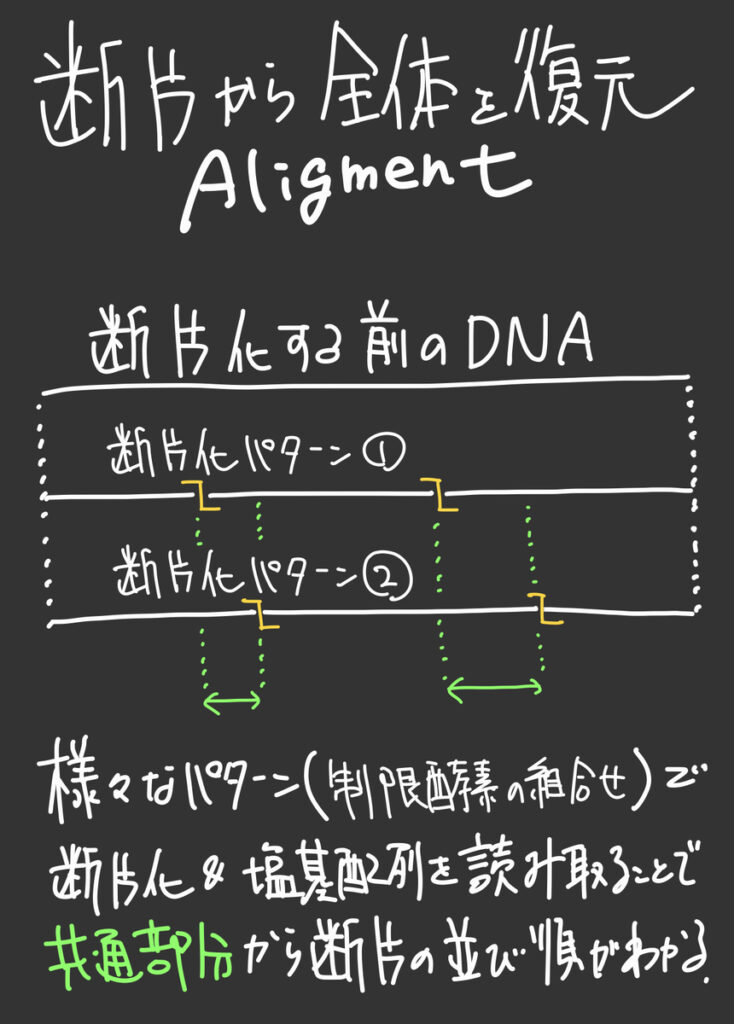
【補足】
- DNAシークエンス(DNAの塩基配列を読み取ること。)
- PCR(ポリメラーゼ連鎖反応。DNAの特定の配列を増幅する方法。)
- DNAポリメラーゼ(DNA合成酵素。新生鎖の3'末端に、鋳型鎖と相補的なヌクレオチドを付け加えていく。プライマーが無ければヌクレオチド差を合成できす、5'末端にヌクレオチドを付加することもできない"わがまま酵素"。)
- ヌクレオチド(リン酸&糖&塩基の結合した分子。DNAやRNAの材料となる。DNAのヌクレオチドは、リン酸&デオキシリボース(糖)に、A,T,G,Cの4種類の塩基のどれかが結合している。)
- dNTP(デオキシリボヌクレオチド三リン酸。4種類のdNTA(dATP,dTTP,dGTP,dCTP)が連結して、DNAのヌクレオチド鎖ができる。)
- ddNTP(ジ・デオキシリボヌクレオチド三リン酸。dNTPでは、糖の3'位置に-OHがあるが、ddNTPは-Hになっている。そのためヌクレオチド鎖の3'末端にddNTPが結合すると、次のdNTP、あるいはddNTPは結合できない。)
- サーマルサイクラー(設定した温度や時間にしたがって、自動で加熱と冷却をくり返してくれる装置。PCRやサンガー法で活躍。)
【参考資料】
- 吉里勝利(2018).『改訂 高等学校 生物基礎』.第一学習社
- 浅島 誠(2019).『改訂 生物基礎』.東京書籍
- 吉里勝利(2018).『スクエア最新図説生物neo』.第一学習社
- 浜島書店編集部(2018).『ニューステージ新生物図表』.浜島書店
- 大森徹(2014).『大学入試の得点源 生物[要点]』.文英堂