【37】サンガー法
サンガー法(ジ・デオキシ法)を使うと、未知のDNAの塩基配列を明らかにできる。
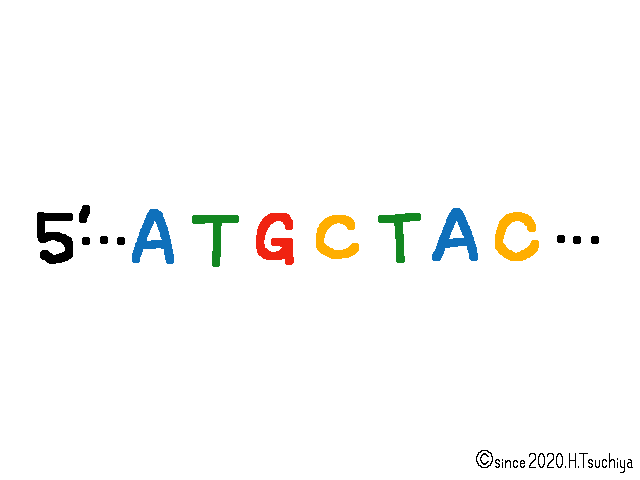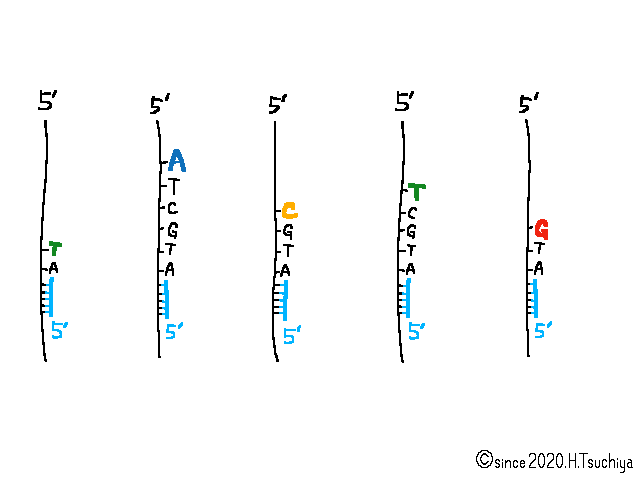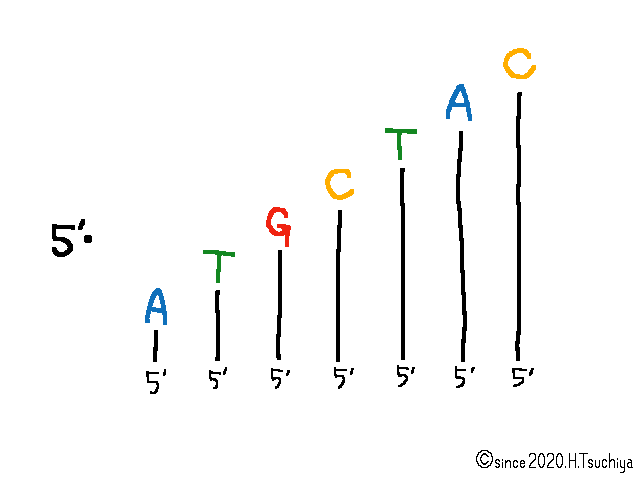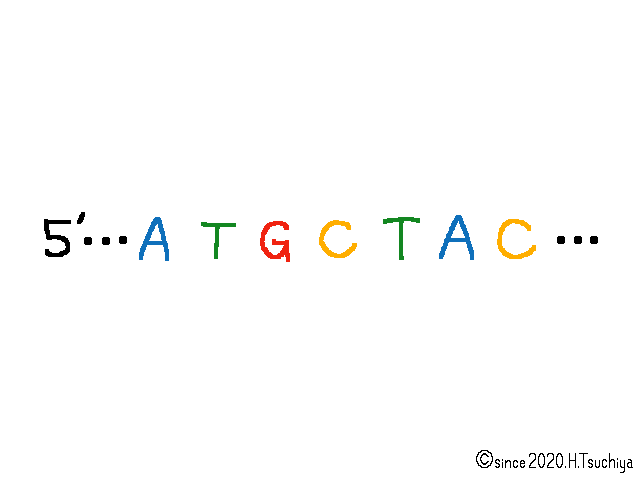
DNA複製の際、DNAポリメラーゼ(DNA合成酵素)は、鋳型鎖と相補的なヌクレオチド鎖(新生鎖)の3‘末端に、新たなヌクレオチドを”脱水縮合”によって付け加える。
このとき、もし新生鎖の3'末端のヌクレオチドの糖が、DNA本来の"デオキシリボース"ではなく、3'位置の-OHが-Hになった”ジ・デオキシリボース”に入れ替っていると、DNAポリメラーゼは新たなヌクレオチドを付け加えることができず、新生鎖の合成はそこで止まってしまう。
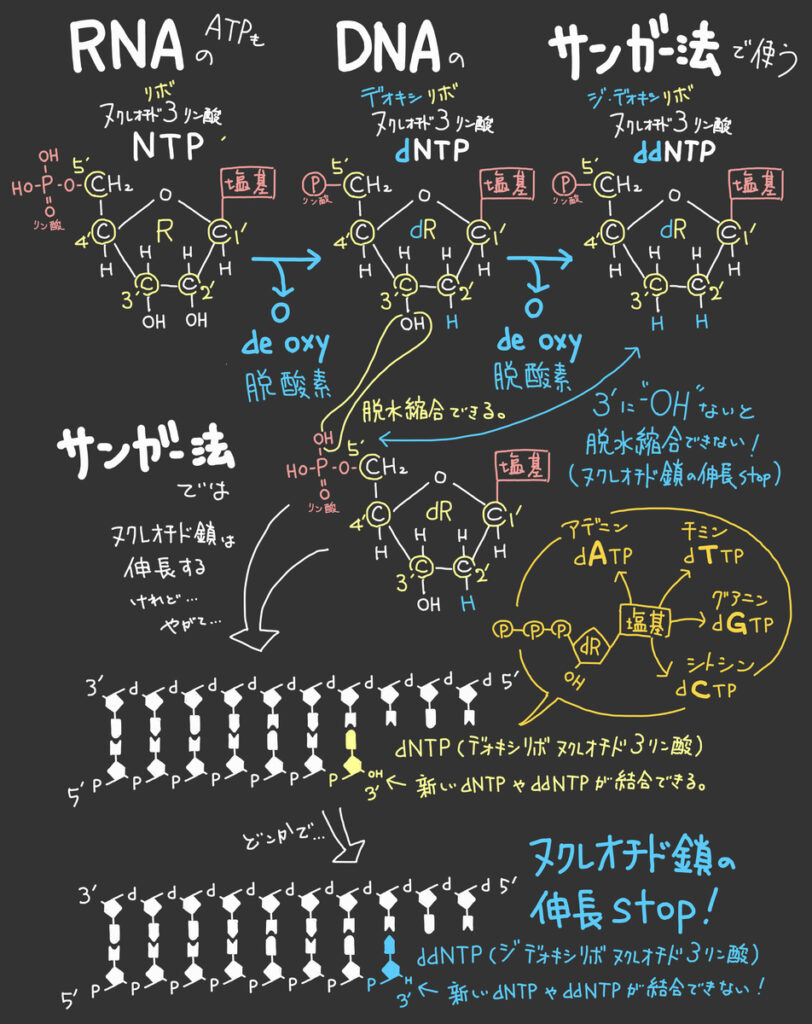
ヌクレオチド鎖の伸長を止めるddNTP(ジ・デオキシリボヌクレオチド三リン酸)を利用すれば、未知のDNAの塩基配列を明らかにすることができる。
- 「塩基配列を明らかにしたいDNA」に、ヌクレオチド鎖の材料になる4種類のdNTP(dATP,dTTP,dGTP,dCTP)、別々の色で蛍光標識した4種類のddNTP(ddATP,ddTTP,ddGTP,ddCTP)、DNAポリメラーゼとプライマーを加える。
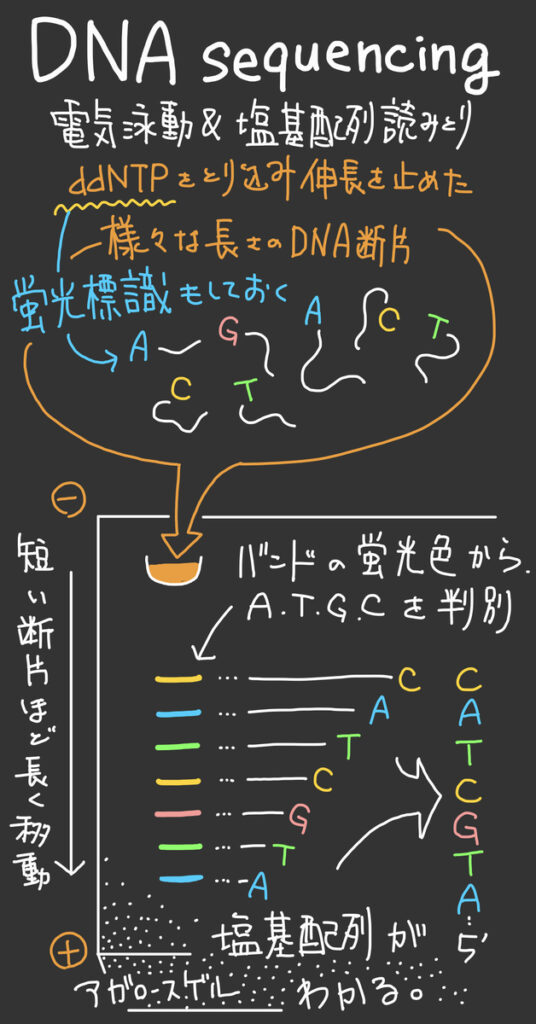
- 1の“混合液”を、サーマルサイクラー(加熱と冷却を自動でくり返す装置)で複製(増幅)する。複製(増幅)のたびに、ddNTPが取込まれて途中で合成が止まった”様々な長さ”のヌクレオチド鎖ができる。
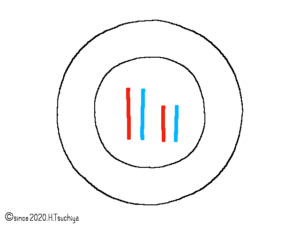
- 2の"様々な長さ"のヌクレオチド鎖を、アガロースゲル電気泳動にかけ、ヌクレオチド鎖の”長さ”の順に並べ替える。
- 3で並べ替えたヌクレオチド鎖(アガロースゲルの上に帯状の”バンド”として見える)の蛍光標識からA,T,G,Cを判別し、5’末端側(最も“長い”距離を泳動した、最も“短い”ヌクレオチド鎖の集まった”バンド”)から、順に塩基配列を読み取る。
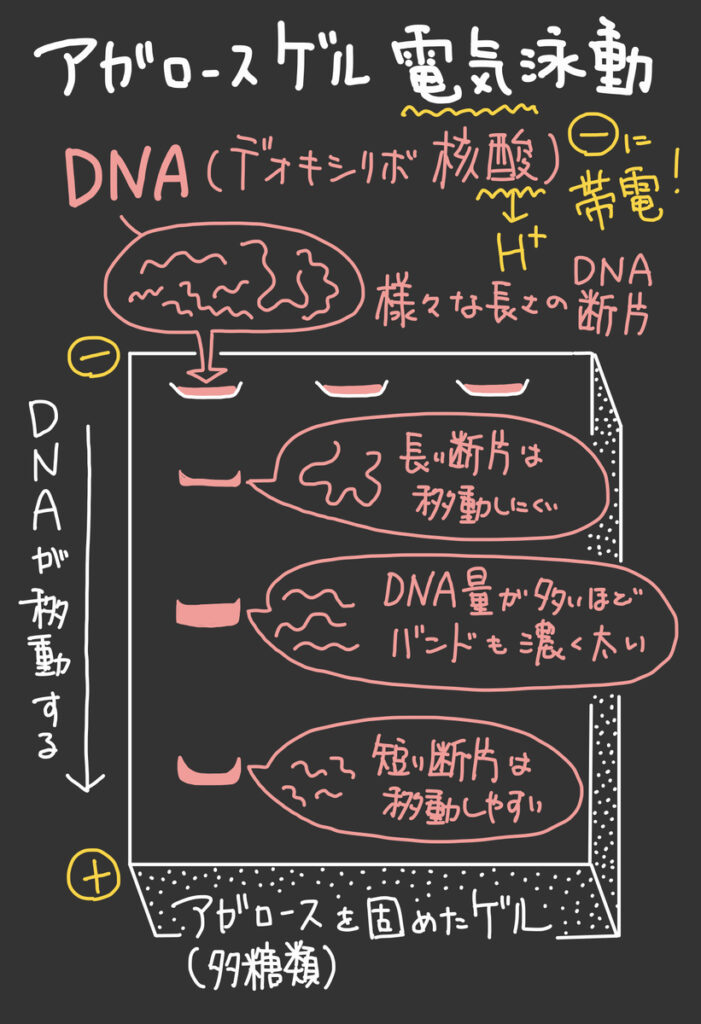
DNA(デオキシリボ核酸)も酸なので、水溶液中では水素イオン(陽イオン)と電離して-に帯電しており、電気泳動によって大きさ(長さ)順に並べ替えることができる。
【補足】
- PCR(ポリメラーゼ連鎖反応。DNAの特定の配列を増幅する方法。)
- ヌクレオチド(リン酸&糖&塩基の結合した分子。DNAやRNAの材料となる。DNAのヌクレオチドは、リン酸&デオキシリボース(糖)に、A,T,G,Cの4種類の塩基のどれかが結合している。)
- dNTP(デオキシリボヌクレオチド三リン酸。4種類のdNTA(dATP,dTTP,dGTP,dCTP)が連結して、DNAのヌクレオチド鎖ができる。)
- ddNTP(ジ・デオキシリボヌクレオチド三リン酸。dNTPでは、糖の3'位置に-OHがあるが、ddNTPは-Hになっている。そのためヌクレオチド鎖の3'末端にddNTPが結合すると、次のdNTP、あるいはddNTPは結合できない。)
- サーマルサイクラー(設定した温度や時間にしたがって、自動で加熱と冷却をくり返してくれる装置。PCRやサンガー法で活躍。)
- DNAポリメラーゼ(DNA合成酵素。新生鎖の3'末端に、鋳型鎖と相補的なヌクレオチドを付け加えていく。プライマーが無ければヌクレオチド差を合成できす、5'末端にヌクレオチドを付加することもできない"わがまま酵素"。)
- アガロース(多数のガラクトースが結合した多糖類。寒天の主成分。アガロースゲル=純度の高い寒天。)
- アガロースゲル電気泳動(アガロースゲルに電流を流すことで、電荷をもつ分子を大きさ順に分離できる。大きい分子ほどゲルに邪魔されて移動しにくく、小さい分子ほど長い距離を移動する。)
- バンド(アガロースゲル電気泳動のさいに現れる帯状の模様。同じ大きさの分子が蓄積して”バンド”として見える。)
- 蛍光色素(特定の波長の光を照射すると、照射した波長とは違う波長の光を発する。)
- シークエンシング(サンガー法などにより、DNAの塩基配列を決定すること。)
- DNAシークエンサー(アガロースゲル電気泳動~塩基配列の読み取りまでを自動で行ってくれる装置。)
【参考資料】
- 吉里勝利(2018).『改訂 高等学校 生物基礎』.第一学習社
- 浅島 誠(2019).『改訂 生物基礎』.東京書籍
- 吉里勝利(2018).『スクエア最新図説生物neo』.第一学習社
- 浜島書店編集部(2018).『ニューステージ新生物図表』.浜島書店
- 大森徹(2014).『大学入試の得点源 生物[要点]』.文英堂